鱼类基因组学及其在抗病育种中的应用
(资料来源:Reviews in Aquaculture)

1 Key Laboratory of Breeding Biotechnology and Sustainable Aquaculture, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan, China
2 BGI Academy of Marine Sciences, BGI Marine, Shenzhen, China
3 College of Life Sciences, University of Chinese Academy of Sciences, Beijing, China
4 Laboratory of Aquatic Genomics, College of Life Sciences and Oceanography, Shenzhen University, Shenzhen, China
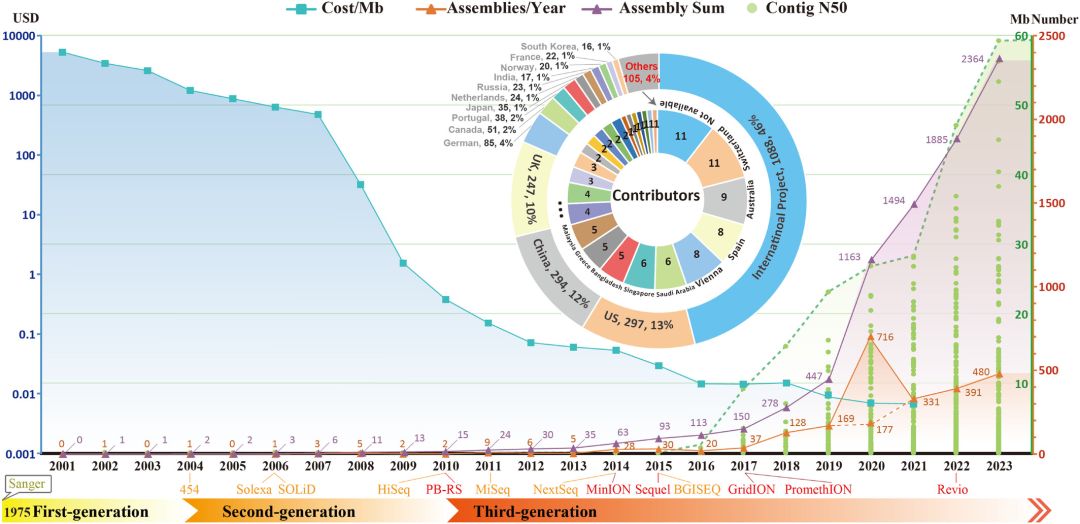
图 基因组学支撑的鱼类抗病育种研究路线图
摘要
近几十年来,全球水产养殖产量一直在攀升,其中约76%来自鱼类。然而,随着水产养殖业发展,病害高发频发、损失日益突出。自2002年报道首个鱼类基因组图谱以来,基因组学方法已成功应用于鱼类抗病育种,培育高抗病力新品种从而减少因病害造成的经济损失。本文就鱼类基因组学及其在鱼类抗病育种中的应用进行综述。本文首先统计分析了所有已报道的鱼类基因组,随后概述了应用在鱼类抗病育种上的基因组辅助育种技术(包括数量性状位点定位、全基因组关联分析、分子标记辅助选择、基因组选择、转基因和基因组编辑等),并总结了可用于鱼类抗病育种的候选遗传标记,最后对该领域目前仍存在的挑战和未来发展方向进行了探讨。总之,本文综述了鱼类基因组学及其相关技术在鱼类抗病育种中的应用与研究进展,为鱼类抗病品种创制提供了新的研究思路。
关键词
水产养殖,育种,抗病,鱼类,基因组学
Abstract
Global aquaculture production has been rising for several decades, with up to 76% of the total production from fish. However, the problem of fish diseases is becoming more and more prominent in today's context of pursuing sustainable aquaculture. Since the first fish genome assembly reported in 2002, genomic approaches have been successfully implemented in fish breeding to enhance disease resistance and reduce economic losses caused by diverse fish diseases. Here, we present a review of the current progress in fish genomics and its application in disease-resistance breeding. First, assembly data for all publicly available fish genomes were curated and statistical analysis of these data were performed. Subsequently, genomics-assisted breeding approaches (including quantitative trait loci mapping, genome-wide association study, marker-assisted selection, genomic selection, gene transfer, and genome editing) that have been applied in practical disease–resistance breeding programs are outlined. In addition, candidate genetic markers that could possibly be utilized in breeding were summarized. Finally, remaining challenges and further directions were discussed. In summary, this review provides insight into fish genomics and genomics-assisted breeding of disease-resistant fish varieties.
keywords
aquaculture, breeding, disease resistance, fish, genomics
作者评述
联合国粮食及农业组织公布的《世界渔业和水产养殖业2024》显示,2022年全球水产养殖总量增至1.309亿吨(鱼类产量增长最快,达58.1%),为保障优质动物蛋白有效供给做出了重要贡献。但随着水产养殖业发展,病害问题日益突出。鱼类病害种类繁多,高发频发常态化,病害防控难度大,基本处于被动防控状态,病害已成为阻碍水产养殖业绿色高质量发展的重要瓶颈之一。针对鱼类重大疾病,相较于传统的化学药物等被动防控手段,研发高效精准的鱼类抗病育种技术、培育抗病新品种可从根本上解决病害防控难题。然而,传统育种技术由于缺少对抗病性状遗传结构特征和抗病分子机制等关键问题的解析,导致育种结果预见性差,存在子代性状分离、育种周期长、难以控制变异方向等问题。
基因组学技术的出现为解决上述问题提供了新的方向。2002年,全球首个鱼类(Takifugu rubripes,红鳍东方鲀)基因组成功破译,截至2024年,已有近700个鱼类物种的全基因组序列得到解析,覆盖了硬骨鱼88%(60/68)的目、46%(197/431)的科,并且包含了全球产量最高的前15个淡水和15个海水/近岸鱼类所有物种,为经济鱼类的高效精准育种奠定基石。我国鱼类基因组研究虽然起步较晚,但现已成为发布鱼类全基因组图谱最多的国家之一。今年正值我国第一篇鱼类(Cynoglossus semilaevis,半滑舌鳎)基因组测序论文发表10周年,我国鱼类基因组研究实现了从跟跑到并跑甚至在个别物种和方向领跑的成功跨越。
这22年不仅见证了鱼类基因组学的发展,还见证了基因组学支撑的相关技术尤其是抗病育种技术的创新和突破。随着基因组学技术的进步,鱼类育种从传统的个体水平、细胞水平和分子水平(通常只有单个或少数几个分子标记)逐渐步入当今的基因组学育种时代。基因组学育种以全基因组解析为基石,以高通量的分子标记(SSR、SNP、InDel等)筛选和优良(如抗病等)性状的遗传解析为核心,通过QTL定位、全基因组关联分析、统计模型算法建立和遗传评估等分析方法,配合分子标记辅助选择、基因组选择、基因编辑等育种技术,实现鱼类高效精准育种和优良性状新品种创制,推动水产种业振兴,支撑鱼类养殖业的绿色可持续发展。
鉴于此,本文对当前所有已公开的鱼类全基因组图谱数据进行了统计和分析,并以此为基础,进一步回顾了基因组学支撑的抗病育种技术的研究现状和进展,归纳了一批候选的抗病分子标记,并深入探讨了鱼类抗病育种目前仍然面临的挑战和未来发展方向。该综述为基于基因组学的鱼类抗病育种研究提供新的视角和启发。
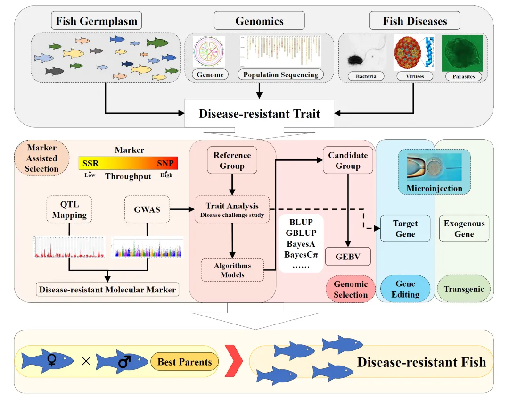
图 基因组学支撑的鱼类抗病育种研究路线图
作者简介
徐镇:博士,中国科学院水生生物研究所研究员,博士生导师,水产品种创制与高效养殖重点实验室主任。主要从事鱼类抗病免疫和病害防控的研究,阐明了病毒入侵感染以及鱼类抗病性状的分子调控机制,为鱼类抗病育种和疫苗研发提供基础。以第一或通讯(含并列)在Science Immunology、Nature Communications等发表论文50余篇。主持国家基金委杰出青年基金、中国科学院先导B项目、生物育种2030“抗病抗逆草鱼培育”课题等。
黄玉:博士,中国科学院水生生物研究所高级工程师。从事鱼类基因组学及分子育种研究,绘制10余种重要经济鱼类全基因组图谱,揭示鱼类特殊性状形成的演化机制,构建可靠的鱼类“生命之树”,为基于基因组的鱼类精准育种提供了基础。以第一(含并列)作者在PNAS、MBE、Cell Discovery等期刊发表论文40余篇,相关成果获科技奖励3项、授权国家发明专利2件。
李泽宇:博士,中国科学院水生生物研究所特别研究助理(博士后)。从事鱼类抗病遗传育种相关研究,在牙鲆中针对迟缓爱德华氏菌病进行了家系选育研究,在大黄鱼中进行了抗内脏白点病遗传基础解析及基因组选择育种研究,目前主要从事草鱼的抗病遗传育种工作。以第一(含并列)作者在Aquaculture、渔业科学进展等杂志发表论文。先后参与国家自然科学基金重点支持项目及面上项目、福建省自然科学基金重点项目等科研项目。
免责申明:我们在分享的同时,十分尊重原创,版权归原创作者所有,如有侵权请联系我们,我们将及时删除